Engenharia genética/Amplificação por PCR do gene GFP de Aequorea victoria: diferenças entre revisões
| [edição não verificada] | [edição não verificada] |
Conteúdo apagado Conteúdo adicionado
m Engenharia Genética:Amplificação por PCR do gene GFP de Aequorea victoria movido para Engenharia genética/Amplificação por PCR do gene GFP de Aequorea victoria: formatação (maiúscula) |
m O ficheiro AgaroseGel.jpg teve de ser removido pois foi apagado no Commons por Killiondude: No source since 4 November 2009 |
||
| Linha 63: | Linha 63: | ||
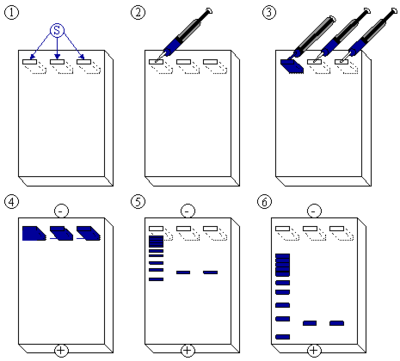
[[Imagem:Agarose-Gelelektrophorese.png|thumb|right|400px|Representação esquemática do processo de electroforese, detalhando as sucessivas etapas. (1) Gel de agarose com três poços. (2) Injecção da solução padrão de DNA marcadora de pesos moleculares, no primeiro poço. (3) Após o padrão estar injectado, injectam-se as amostras no segundo e terceiro poços. (4) Aplica-se uma corrente eléctrica que provoca a migração do DNA para o pólo positivo, devido à carga negativa conferida pelo esqueleto fosfatado. (5) As cadeias de DNA pequenas migram mais rapidamente, por outro lado, as maiores migram mais lentamente através do gel; normalmente, durante este processo, o DNA não é visível, por isso adiciona-se ao DNA um corante ou marcador para evitar que o DNA migre para fora do gel. (6) Adicionar o corante ou marcador também ao padrão de DNA.]] |
[[Imagem:Agarose-Gelelektrophorese.png|thumb|right|400px|Representação esquemática do processo de electroforese, detalhando as sucessivas etapas. (1) Gel de agarose com três poços. (2) Injecção da solução padrão de DNA marcadora de pesos moleculares, no primeiro poço. (3) Após o padrão estar injectado, injectam-se as amostras no segundo e terceiro poços. (4) Aplica-se uma corrente eléctrica que provoca a migração do DNA para o pólo positivo, devido à carga negativa conferida pelo esqueleto fosfatado. (5) As cadeias de DNA pequenas migram mais rapidamente, por outro lado, as maiores migram mais lentamente através do gel; normalmente, durante este processo, o DNA não é visível, por isso adiciona-se ao DNA um corante ou marcador para evitar que o DNA migre para fora do gel. (6) Adicionar o corante ou marcador também ao padrão de DNA.]] |
||
[[Imagem:AgaroseGel.jpg|thumb|centre|250px|Gel de agarose preparado para análise de DNA. O primeiro poço contém um padrão de DNA para determinação de pesos moleculares; os quatro poços seguintes mostram fragmentos de DNA com variados tamanhos, que estão presentes em algumas mas não em todas as amostras.]] |
|||
Revisão das 06h29min de 17 de novembro de 2009
acima: ÍNDICE
Preparação das soluções para PCR

- Fazer uma diluição de 50X, em 2 tubos eppendorf marcados, fazendo 2 diluições sucessivas de 10X a partir de 100 μL de Taq e 900 μL do tampão de diluição 1X e depois 5X (200 μL → 800 μL);
- Fazer 3 diluições sucessivas de 2X em 3 tubos eppendorf marcados, a partir de 500 μL da diluição de 1000X e 500 μL de tampão de diluição 1X;
- Colocar 20 μL das diluições em diferentes tubos de PCR: 10x, 50x, 100x, 200x e 400x (5 tubos);
- Adicionar 20 μL de PCR MasterMix a cada tubo de PCR;
- Colocar os tubos em gelo;

- Depois de agitadas no vortex, as misturas são sujeitas a PCR de acordo com as condições especificadas na
tabela abaixo.
Condições do PCR
| Temperatura | Duração | Fase |
| 94 ºC | 120 seg | Desnaturação inicial |
| 94 ºC | 20 seg | Desnaturação |
| 40 ºC | 20 seg | Emparelhamento |
| 72 ºC | Taq = 20 seg e Pfu= 1 min 45 seg | Elongamento |
| 72 ºC | 120 seg | Elongamento final |
Esta sequência é repetida 30 vezes.
Análise Electroforética dos produtos de PCR
- Misturar 5 μL de produto de PCR com 5 μL de tampão de aplicação;
- Aplicar as amostras num gel de agarose 0,8%;
- O instrutor efectua a electroforese com as amostras correspondentes aos produtos de PCR de referência;
- Correr o gel, durante 20 minutos, a 220 volts;
- Fotografar o gel.

Actividades propostas
- Comparar o rendimento do PCR obtido com o da Taq polimerase comercial.
- Calcular o número de unidades na preparação.
