Engenharia genética/Amplificação por PCR do gene GFP de Aequorea victoria
Preparação das soluções para PCR
[editar | editar código-fonte]
- Fazer uma diluição de 50X, em 2 tubos eppendorf marcados, fazendo 2 diluições sucessivas de 10X a partir de 100 μL de Taq e 900 μL do tampão de diluição 1X e depois 5X (200 μL → 800 μL);
- Fazer 3 diluições sucessivas de 2X em 3 tubos eppendorf marcados, a partir de 500 μL da diluição de 1000X e 500 μL de tampão de diluição 1X;
- Colocar 20 μL das diluições em diferentes tubos de PCR: 10x, 50x, 100x, 200x e 400x (5 tubos);
- Adicionar 20 μL de PCR MasterMix a cada tubo de PCR;
- Colocar os tubos em gelo;

- Depois de agitadas no vortex, as misturas são sujeitas a PCR de acordo com as condições especificadas na
tabela abaixo.
Condições do PCR
[editar | editar código-fonte]
| Temperatura | Duração | Fase |
| 94 °C | 120 seg | Desnaturação inicial |
| 94 °C | 20 seg | Desnaturação |
| 40 °C | 20 seg | Emparelhamento |
| 72 °C | Taq = 20 seg e Pfu= 1 min 45 seg | Elongamento |
| 72 °C | 120 seg | Elongamento final |
Esta sequência é repetida 30 vezes.
Análise Electroforética dos produtos de PCR
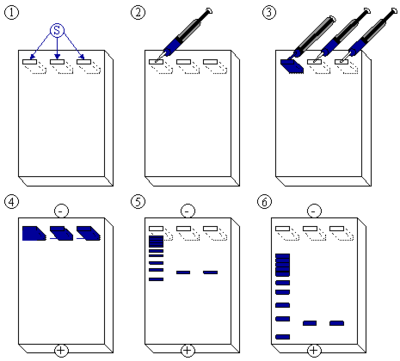
[editar | editar código-fonte]- Misturar 5 μL de produto de PCR com 5 μL de tampão de aplicação;
- Aplicar as amostras num gel de agarose 0,8%;
- O instrutor efetua a eletroforese com as amostras correspondentes aos produtos de PCR de referência;
- Correr o gel, durante 20 minutos, a 220 volts;
- Fotografar o gel.

Atividades propostas
[editar | editar código-fonte]- Comparar o rendimento do PCR obtido com o da Taq polimerase comercial.
- Calcular o número de unidades na preparação.
